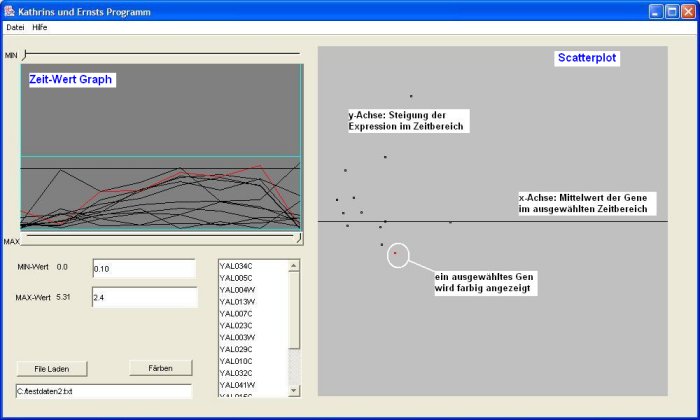
Unsere Implementierung
Für die Umsetzung der Ideen des Papers haben wir Java 1.4.2_03 mit Hilfe
von Jbuilder 9 verwendet. Mit unserer Implementierung, die an das Paper "Coordinated
Graph and Scatter-Plot Views for the Visual Exploration of
Microarray Time-Series Data" angelehnt ist, haben wir versucht, die Grundfunktionalitäten
zu implementieren und ein intuitives und voll funktionsfähiges System zur
Visualisierung von Microarray Daten programmieren.
Die zu visualisierenden Daten werden aus einem Datenfile im txt-Format eingelesen.
Übersicht:

Funktionalitäten:
Der User kann messbare Abfragen mit separaten Zeitperiode und Bedingungskomponenten visuell formulieren und modifizieren.Grundsätzlich werden die visuellen Abfragen durch die Kombination aus traditionellen Zeitdiagramm Repräsentationen der Daten mit einer ergänzenden Scatterplot Repräsentation einer definierten Zeitperiode unterstützt.
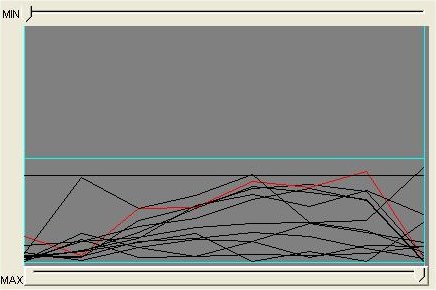
Zeit/Wert Graph
Durch die Slider kann ein Zeitbereich ausgewählt werden. Mit den eingegebenen Werten (siehe unten) wird ausgewählt, welche Linien angezeigt werden.
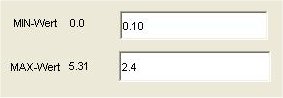
Wertebereich auswählen
Hier kann ausgewählt werden, wie hoch die Genexpression der angezeigten Daten maximal sein soll. Diese Abfrage wird im Zeit/Wert Graphen durch die türkisen Linien dargestellt. Es werden nur noch die Linien angezeigt, die der Auswahl entsprechen
Einzelne Gene auswählen
Werden einzelne Gene ausgewählt, so werden sie automatisch in beiden Graphen farbig dargestellt.
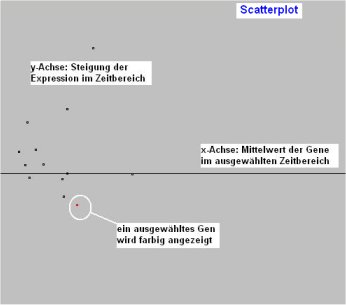
Scatterplot
Auf der Y-Achse ist die Veränderung eingetragen und auf der
X-Achse die durchschnittlichen Werte. Die druchschnittlichen Werte sind für
jedes einzelne Gen für die Zeitperiode berechnet worden und nicht zu verwechseln
mit dem Mittelwert aller Gene in dem Zeitabschnitt.