Koordinierte Graph und Scatter-plot Ansichten
für die visuelle Exploration von Microarray
Zeitreihendaten
2.
Evaluierung von bestehenden Systemen
Anforderung
1: Gen aktiv/ nicht aktiv?
Anforderung
2: steigende oder nachlassende Expression?
Anforderung
3: Ähnlichkeitsidentifikation
Anforderung
4: existierenden funktionellen Klassifikationen
Anforderung
5: Exportieren von Ergebnissen
Einschränkungen
existierender Techniken
3.3.
Koordination der Ansichten
Abstract
Microarrays sind eine relativ neue Datenerfassungs (gewinnungs-) Technologie mit hoher Durchsatzleistung. Typische Prozeduren von Microarray Experimenten sind Zeitablauf Experimenten, deren Ergebnisse Zeitreihen sind. Die Analyse der Ergebnisse hat das Potential, einen wichtigen Einfluss auf Diagnose, Behandlung und Krankheitsvorbeugung zu haben. Existierende Informationsvisualisierungstechniken machen die Daten leichter handhabbar, eine Anforderungsanalyse hat jedoch zahlreiche Einschränkungen ergeben. Die wichtigste Erkenntnis war, dass es Usern nicht möglich war, Wertveränderungen während eines bestimmten Zeitabschnittes (über den Zeitabschnitt) zu finden und zu quantifizieren. Das Paper beschreibt eine neue Technik, die diese Funktionalität zur Verfügung stellt, indem der User messbare Abfragen mit separaten Zeitperiode und Bedingungskomponenten visuell formulieren und modifizieren kann, d.h. mittels einer intuitiven direkt manipulierbaren Schnittstelle. Die visuellen Abfragen werden unterstützt durch die Kombination aus traditionellen Zeitdiagramm Repräsentationen der Daten mit einer ergänzenden Scatterplot Repräsentation einer definierten Zeitperiode. Die verschiedenen Ansichten sind aufeinander abgestimmt, so dass der User Abfragen formulieren und modifizieren kann, mit schnell umschaltender Anzeige der Ergebnisresultate im traditionellen Wert gegen Zeit Diagrammformat. Die Technik gibt den Usern eine einzigartige, direkt animierte Ansicht von Microarray Zeitreihen, die ihnen erlaubt, Muster über die Zeit für das gesamte Datenset oder für ausgewählte Subsets zu explorieren. Die Kombination aus einem messbaren Abfragemechanismus und intuitiven, direkt manipulierbaren Interface ermöglich es Biologen ihre eigenen Abfragen zu formulieren und die Resultate zu quantifizieren. Anfängliche Testungen der Tools zeigen, dass es User einfach finden sich an die unterschiedlichen Ansichten zu gewöhnen und wichtige Eigenschaften in ihren Daten identifizieren können.
Definition Microarrays
Ein Microarray (DNS-Chips) ist ein Glasträger, auf dem kurze DNA-Sequenzen
punktgenau aufgebracht sind.
Die Microarray-Technik erlaubt eine umfassende
Analyse der Genexpression auf mRNA-
und Proteinebene: Da mit einer einzigen Untersuchung mehrere
1 000 Gene gleichzeitig analysiert werden können, erhält man
Momentaufnahmen komplexer Expressionsmuster.
((Das Realisieren der Information, die in der DNA eines Gens gespeichert ist, nennt man Genexpression: Gen --> Protein(e) -> Merkmal.))
1. Einleitung
Zeitreihen sind eine Sequenz von Beobachtungen, die nach einem Zeitparameter sortiert sind. Die Beobachtungen werden typischerweise periodisch gemessen und stellen die beobachtete Realisation eines Ereignisses dar. Es kommt oft vor, dass eine große Sammlung von mit einander in Beziehung stehenden Zeitreihen zusammen eine beobachtete Realisation eines zugrunde liegenden Systems darstellt. Beispiele solcher Zeitreihen: wirtschaftliche Daten, Aktiendaten, medizinische und Microarray Daten.
Die Studie arbeitet mit Microarray Zeitablauf Experimenten, deren Ergebnisse Zeitreihen Daten sind. Microarrays sind eine relativ neue Datenerfassungs (gewinnungs-) technologie mit hoher Durchsatzleistung. Der Ursprung der Zeitreihen sind Gene und die aufgezeichneten Daten sind Gen Expressionen.
Individuelle Zeitreihen werden öfter als Expressionsmuster oder -profile bezeichnet.
Mit der Microarray Technologie werden Unmengen von Daten produziert, welche wichtig sich in der medizinischen Forschung, die Datenanalyse und –bearbeitung stellt jedoch ein großes Problem bei der Nutzung dieser Technologie dar.
Die 2 Hauptansätze für die Visualisierung von Microarray Daten sind Clustering Techniken, die Zeitreihen gemäß vordefinierten Ähnlichkeitsmaßen gruppieren und visuelle Abfragemethoden, die dem User erlauben, die Daten interaktiv zu exploriere. Übliche Datenaufbereitung für die Visualisierung sind Normalisierung und Skalierungsänderung.
Der verbreitetste Ansatz zur Visualisierung von Microarray Zeitreihen ist Clustering. Die Gründe dafür sind einleuchtend. Durch Clustering von ähnlichen Elementen kann der Datananalyst zwischen Gruppen von Elementen verallgemeinern und muss nicht einzelne Elemente beachten. Durch das Clustering kommt man leichter mit Daten un deren große Menge zurecht. (Microarray Experimente zeichnen typischerweise die Expression von ca. 6000 Genen auf).
Die erste Stufe des Clustering ist die Erzeugung einer Ähnlichkeitsmatrix. In dieser stellen die Zeilen und Spalten die gesamte Liste der Gene dar- die Zellen spiegeln die Ähnlichkeit zwischen Zeitreihen, die nach einem vordefinierten Ähnlichkeitsmaß berechnet wurden. Die direkte Visualisierung ist ein Raster, in dem die Helligkeit der Zellen den Grad der Ähnlichkeit ausdrücken.
Die üblichste Clusteringmethode basiert auf dem Resultat von agglomerativen hierarchischem Clustering, welches die Ähnlichkeitsmatrix verwendet um einen binäre Baum zu produzieren, der als Dendrogramm bekannt ist.

In der üblichen Visualisierung eines Dendrogramms repräsentieren die Endpunkte der äußeren Zweige Gene und die Gruppierung nach Ähnlichkeit wird durch die gemeinsamen Zweige in der Baumstruktur dargestellt. In der Darstellung sind Gen Expressionsmuster farbkodiert und vertikal neben ihren zugehörigen Gen Knoten aufgestapelt. Dieser Teil der Anzeige ist bekannt als Farbmosaik.
Eine andere Alternative Resultate von agglomerativen hierarchischem Clustering darzustellen: Veränderung von parallelen Koordinaten wird verwendet um Aggregationsinformation für die Cluster zu vermitteln. Der Display ist hochauflösend und erlaubt dem User die Dendrogrammstruktur zu navigieren, bis die erwünschte Fokusregion und Detaillierungsgrad erreicht ist.
Clustering ist geeignet bestimmte natürliche Gruppierungen aufzudecken und eine globale Sicht der Daten zu bieten, jedoch ist es stark subjektiv und die Signifikanz von dem was man sieht ist nicht quantifiziert. Desweiteren können einzelne Zeitreihen mehrere interessante Merkmale enthalten, die nicht in einem einzelnen Ähnlichkeitsmaß berücksichtigt werden können, auf dass das Clustering letzlich beruht. Die Abstraktion der Daten, aufgrund von Ähnlichkeit führt zu einem Verlust von wichtigen Informationen. Zb. wenn ein biologisch wichtiges Merkmal nur in einem begrenzten Zeitabschnitt des Gesamtzeitrahmens auftritt, kann es verwässert werden von der Repräsentation von weniger wesentlichen Merkmalen die im restlichen Zeitrahmen auftauchen.
Die primäre Alternative zu Clustering der Microarray Zeitreihen ist visuelle Erkundung (Querying).
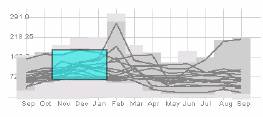
Visuelle Abfragetools zeigen einen Überblick über die der Daten, in Form von überlagerten Wert gegen Zeit Graphen. Durch klicken und ziehen kann man visuelle Abfragen formulieren und es werden Boxen (Zellen) obenauf über den Überblick gezeichnet um einen akzeptablen Wertebereich über eine gewisse Zeitperiode zu spezifizieren. Sukzessive Abfragen filtern die Daten, so dass der anfängliche Überblick ersetzt wird durch überlagerte Wert/Zeit [in Abhängigkeit, gegen] von Graphen eines gefilterten Teilsets. Die TimeSearcher Technik erweitert dieses Basismodel , indem es dem User erlabt die Abfragen zu modifizieren, in dem er klickt und Kanten oder Ecken der visuellen Abfragerepräsentationen zieht (verschiebt), während ein schnell umkehrbares Update der Abfrageresultate gezeigt wird.
Der zentrale Vorteil von visuellen Abfragen gegenüber Clustering ist dass Abfragen messbar sind und es leicht für den User ist seine Resultate zu quantifizieren. Ein weiterer Vorteil ist, das Abfragen eingeschränkt werden können auf spezifizierte Zeitperioden. So können auch biologisch wichtige Merkmale, die nur in einem begrenzten Zeitabschnitt des Gesamtzeitrahmens auftreten, identifiziert werden.
Der Nachteil bestehender visuellen Abfragetechniken ist, dass der User weniger effektiven Überblick über die Daten haben wird als bei Clustering. Der überlagerte Wert gegen Zeit Graph Repräsentation der Zeitreihen kann eine große Anzahl voneinander verschiedenen Zeitreihen nicht mehr in Einklang bringen, ohne dass einzelne Zeitreihen unleserlich aufgrund der überkreuzenden Linien werden. Die Repräsentation wie in Abb. 2 ist nur effektiv im Aufdecken von extremen Werten und dem Ausmaß von abseits gelegenen Werten, zu individuellen Zeitpunkten.
Kommerzielle Microarray Tools kombinieren oft Clustering und visuelle Abfragesichten der Daten in verschiedenen aufeinander abgestimmten Fenstern. Trotzdem erfüllen diese Tools nicht alle Benutzerbedürfnisse.
2. Evaluierung von bestehenden Systemen
Die Forscher des Papers haben eine detaillierte Bedürfnisanalyse durchgeführt und es resultierten auf hoher Ebene folgende Aufgaben, die ein Microarray Zeitreihen Tool bewältigen sollte:
- Zeige welche Gene aktiv sind und welche nicht in einer festgelegten Zeitspanne des Gesamtzeitrahmens.
- Zeige, welche Gene eine steigende oder nachlassende Expression in einer gewissen Zeitspanne haben.
- Ermögliche die Identifikation ähnlicher Expressionsmuster.
- Bringe aufgedeckte (offenbarte) Merkmale mit existierenden funktionellen Klassifikationen in Bezug.
- Erlaube das Exportieren von Ergebnissen.
Anforderung 1: Gen aktiv/ nicht aktiv?
Begriff ist mehrdeutig und vom Grad der Genexpression abhängig. Da Gene unterschiedlich sensitiv sind, ist es auch unterschiedlich, bei welchem Grad der Genexpression ein Gen aktiv ist. Ein üblicher Ansatz um zu bestimmen, ob Gen zu einem bestimmten Zeitpunkt aktiv ist, ist die Reskalierung individueller Zeitreihen abhängig von deren Maximalwert. D.h. es wird ein Prozentwert angegeben, dadurch sind Werte verschiedener Zeitreihen vergleichbar. Das Problem dabei ist, dass wenn die Genexpression die gesamte Zeit hindurch sehr niedrig ist, würde eine unrelevante Variation dazuführen, dass das Gen aktiv scheint, obwohl es die ganze Zeit ausgeschaltet ist. Daher ist es auch notwendig, die reskalierten sowie die absoluten Werte über die Zeitperiode darzustellen um feststellen zu können, ob das Gen aktiv ist oder nicht.
Anforderung 2: steigende oder nachlassende Expression?
Um die Veränderung des Wertes über die Zeit zu berechnen ist es nötig, den Anfangswert vom Endwert abzuziehen.
Damit der User die Aussagekraft einer Wertveränderung beurteilen kann ist es erneut nötig die Zeitreihen zu reskalieren. Die reskalierten Werte sind Prozentwerte relativ zur maximalen Veränderung. Diese Methode macht es möglich Anstieg oder Abfall in Werten über Zeit zu vergleichen, es ist jedoch nicht möglich, einen Anstieg mit einem Abfall zu vergleichen. Damit es möglich ist, dass Wertanstieg oder -abfall zu vergleichen, ist es notwendig log fold change in Betracht zu ziehen/ zu berücksichtigen, welche einen x Faktor Veränderung zu einem das gegenteiligen Äquivalent einer 1/x Faktor Veränderung macht. Dazu ist eine Log Reskalierung nötig. Bei microarray Daten werden log skalierte Daten oft als Indikator verwendet, zu welchem Grad ein Gen aktiviert/deaktiviert ist, aber der zentrale Vorteil Microarray Daten zu log skalieren ist Veränderungen der Aktivierung zu berichten (in Bezug zu bringen). Ein nützliches Merkmal der Log skalierung ist, die Veränderung zwischen logskalierten Werten ist gleichzusetzen mit dem log fold Veränderung zwischen Werten. Aus denselben Gründen wie im letzen Punkt ist es nötig, beide Sichten zu haben- die log skalierten und die absoluten Werte.
Anforderung 3: Ähnlichkeitsidentifikation
Dem User sollte es möglich sein, ähnliche Expressionsmuster zu finden. Interessierende Merkmale sind der Grad der Expression und die Veränderung der Expression über die Zeit. Daher ist es logisch das Ähnlichkeitsmaße diese Merkmale reflektieren sollen. Ähnlich könnten zb. Expressionsmuster sein, wo die Expression am Start ansteigt und dann nachlässt, andere wo die Expression später ansteigt und hoch bleibt. Also ist es notwendig, dass Gruppierung für die verschiedenen Wert und Veränderungsmerkmale über verschiedene Zeitperioden möglich ist.
Anforderung 4: existierenden funktionellen Klassifikationen
Existierende funktionelle Klassifikationen sollten sich in der Visualisierung wiederspiegeln. Diese Option ist bisher meist nicht möglich, obwohl interessierende Merkmale der Daten anhand der funktionellen Klassifikation eingeteilt werden.
Anforderung 5: Exportieren von Ergebnissen
Abfragen und Ergebnisse sowie Teile daraus müssen exportierbar sein um sie unter Mitarbeitern auszutauschen.
Einschränkungen existierender Techniken
Clustering Techniken: Anforderung 3 (Ähnlichkeit) erfüllt, jedoch können keine Merkmale aufgedeckt werden, die nur eine kurze Zeitperiode existieren. Daher Anforderung 1 und 2 nicht erfüllt. Die subjektive Natur der Anzeigen macht auch Anforderung 5 (Export) nicht möglich, da Anzeigen/Resultate nicht zum Austausch geeignet sind. Erfordernis 4 (funktionelle Klassen) kann erfüllt werden, wenn diese das Display überlagern.
Visuelle Abfragen der Zeitreihendaten: Zeitreihen können anhand eines festgesetzten Bereiches von Werten über eine festgesetzte Zeitperiode gruppiert werden: Das ermöglicht dem User herauszufinden, welche Gene aktiv/inaktiv sind. Wenn der User mehrere Wertbereiche für aufeinander folgende Zeitperioden spezifiziert, wird die Visualisierung die Wertveränderung über die Zeit wiederspiegeln. Trotzdem ist der tatsächliche Grad der Veränderung nicht quantifizierbar. Erweiterte Parallele Koordinaten erlauben Abfragen, bei den man einen erlaubten Gradienten zwischen benachbarten Dimensionen festsetzten kann. Gradienten zwischen nicht benachbarten Dimensionen abzufragen ist nicht möglich, da die Dimensionen erneut geordnet werden müssten, was für Zeitreihendaten nicht möglich ist, da die Reihenfolge der Dimensionen eine fundamentale Eigenschaft ist. Anforderung 3 (Ähnlichkeit) ist teilweise erfüllt, User kann ähnliche Zeitreihen finden, bei denen Ähnlichkeit auf verschiedene Wert und Veränderungsbedingungen über aufeinander folgende Zeitperioden basiert, Veränderung kann jedoch nicht in ausreichender Weise für nicht anschließende Zeitperioden quantifiziert werden. Anforderung 4 (funktionelle Klassen) kann nicht erfüllt werden, indem man die Klassen darüber legt, da einzelne Zeitreihen nicht getrennt voneinander sind wegen dem Problem der kreuzenden Linien.
Reskalierung: Anforderung 1 und 2 benötige beide, dass Werte sowie Veränderungen für originale und für reskalierte Zeitreihen angezeigt werden. Oftmals wird es oft so gesehen, dass die Reskalierung der Visualisierung vorausgehen muss. In keinem Tool gibt es die Möglichkeit, während dem Analyse Prozess zwischen reskalierten Versionen der Daten hin und her zuwechseln.
Visuelle Abfragen: Während Clustering Techniken gut sind um einen Überblick über Microarray Daten zu bekommen, erlauben visuelle Abfragemechanismen dem User Ergebnisse präziser einzuschränken und zu quantifizieren.
Da die User die Bedürfnisse hatten, die Ergebnisse zu quantifizieren, so dass Abfragen und Resultate geteilt und wieder verwendet werden konnten, wurde der Ansatz der visuellen Abfragen präferiert. Die wichtigste Einschränkung bestehender visueller Abfragetechniken war, das Abfragen nur einen festgesetzten Wertebereich über eine Zeitperiode und nicht einen festgesetzten Veränderung s bereich spezifizieren konnten.
Die bestehenden möglichen Abfragen:
![]()
v ist der Zeitreihen Wert zum Zeitpunkt t, Qv ist der spezifizierte Wertebereich über Zeitperiode Qt
Der User benötigt zusätzlich diese Abfragen:
![]()
![]() ist die erlaubte Wertveränderung.
ist die erlaubte Wertveränderung.
Der User benötigt auch reskalierte Ansichten der Daten, daher sind auch folgende 2 Abfragen nötig:
![]()
![]()
QPM ist der Bereich der Prozentsatz in Bezug auf den reskalierten maximalen Wert, PM ist der Prozentsatz gg. dem maximalen Wert ,
![]() ist die erlaubte Veränderung in logskalierten Daten, die auch Fold
Veränderung genannt wird. LS bedeutet logskalierter
Wert.
ist die erlaubte Veränderung in logskalierten Daten, die auch Fold
Veränderung genannt wird. LS bedeutet logskalierter
Wert.
Jede der Abfragetypen kann in einen Bedingungskomponente, einen Zeitbezug und Reskalierung aufgegliedert werden.
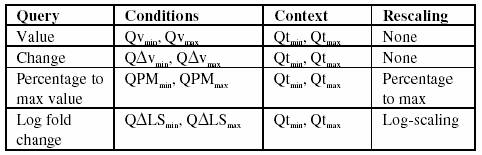
In bestehenden Tools sind nur Wert Abfragen bzw. Prozente im bezug auf den maximalen Wert, falls die Daten vorher reskaliert wurden.
Um alle diese Abfragen zu unterstützen, war es nötig, eine neue Visualisierungstechnik zu entwickeln.
3. Der Ansatz des Papers
Da unsere User die Daten studieren und analysieren wollen und Ergebnisse oft zu weiteren Analysen führen, ist es einfach unser Tool als exploratives System zu klassifizieren. Für unser User, die sich zwar am Computer weniger gut, umso mehr jedoch in ihrem Fachgebiet auskennen, scheint es am geeignetsten, wenn sie die Daten direkt in der Repräsentation manipulieren können.
Diese direkten Abfragen lassen sich jedoch nicht direkt mit Parametern quantifizieren, daher sollte es auch visuelle Abfragen geben bei denen dann Abfragekomponenten direkt über der Datenvisualisierung gelegt werde. Folgende Parameter müssen messbar repräsentiert werden in der Visualisierung für die visuellen Abfragen:
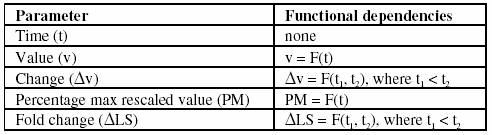
Für Veränderung und Fold Veränderung werden Anfangs und Endzeitpunkt der Veränderung benötigt.
Nach Shneidermans’
Datentyp Taxonomie wurden die Datentypen
kategorisiert, die für die Visualisierung benötigt wurden: die Zeitreihendaten
konnten als temporäre Daten klassifiziert werden. Jedoch bei visuellen Abfragen
von Veränderung und Fold Veränderung, was
Eigenschaften der Daten sind, die von einer Zeitperiode abgeleitet wurden, sind
die Daten zweidimensional, mit den Dimensionen ![]() und
und ![]() für jede Zeitperiode. Für Zeitperioden mit n
Beobachtungen, gibt es 2n mögliche Zeitperioden. Daher ist es
natürlich unmöglich alle Veränderungen zu zeigen. Um messbare Ansichten aller Parameter zu
zeigen, sind mehrere Ansichten nötig. Ein Überblick, der die Daten abhängig von
einem einzigen Zeitpunkt zeigt (zeitlicher Aspekt der Daten) und eine
Detailansicht, die Dateneigenschaften abhängig von der Zeitperiode zeigt (2 dimensionale
Unterdatensätze für ausgewählte Zeitperioden).
für jede Zeitperiode. Für Zeitperioden mit n
Beobachtungen, gibt es 2n mögliche Zeitperioden. Daher ist es
natürlich unmöglich alle Veränderungen zu zeigen. Um messbare Ansichten aller Parameter zu
zeigen, sind mehrere Ansichten nötig. Ein Überblick, der die Daten abhängig von
einem einzigen Zeitpunkt zeigt (zeitlicher Aspekt der Daten) und eine
Detailansicht, die Dateneigenschaften abhängig von der Zeitperiode zeigt (2 dimensionale
Unterdatensätze für ausgewählte Zeitperioden).
Da Interface der Microarray Zeitreihen beinhaltet folgende Komponenten:
- Graphische Übersicht für die Zeitperioden Spezifikation
- Eine Scatterplot Detailansicht einer spezifizierten Zeitperiode
- Eine textuelle Liste aller Gen Namen
- Eine Reihe von Bereichsschiebebalken (range sliders) für Abfragenaufbau (komposition)
- Eine kombinierte Steuerfläche (panel) für Abfragen und eine Steuerfläche zum Aufdecken vordefinierter Gruppen
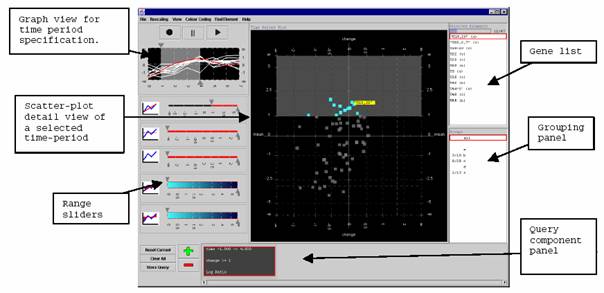
3.1 Überblick
Die erste logische Stufe im Design eines Visualisierungstools ist der Überblick. Der User soll ein Gefühl für das gesamte Datenset bekommen, und er soll die allgemeine Streuung der Daten beurteilen können. Außerdem bietet der Überblick den Kontext für zusätzliche Detailansichten. Im Überblick werden tatsächliche Daten und nicht abgeleitete Qualitäten, die bei der Datenmanipulation helfen, dargestellt.
Grundsätzliche Strategien, Zeitdaten zu visualisieren sind statische und animierte Repräsentationen. In statischen Repräsentationen wird Zeit als fixierte Achse des gesamten Displays dargestellt. In Animationen kann der User alle festgelegten Datenattribute beobachten zu jedem Zeitpunkt beobachten, während die Zeit konstant vergeht. Animationen sind aber wenig üblich, da es schwierig für den User ist zu vergleichen.
Es wurde ganz einfach die graphische Repräsentation gewählt, bei der Zeit auf der x-Achse und die Werte auf der y-Achse abgebildet sind.
Dich Zeit und Wert Achsen erlauben dem User, Zeiten und Werte auszuwählen, d.h. Wertebereiche zu spezifizieren (auch Zeitperioden für Veränderungen und Fold Veränderungen)
Die Auswahl der Zeitperiode ist primär, da alle zuvor beschriebenen Abfragen einen Zeitabschnittskontext besitzen. Da es zahlreiche Abfragen gibt, bei denen die Zeitkomponente wichtig ist und der User oftmals einen Zeitabschnitt definieren will, jedoch keinen Wertebereich, wurde die mögliche Interaktion mit dem Zeit/Wert Diagramm darauf begrenzt.
Diese Funktion wurde mit einem Zeitschieber realisiert. Der innere Raum des Schiebers wird verwendet um eine graphische Wert/Zeit Repräsentation unserer Zeitreihendaten darzustellen. Eine kleine Änderung des grundlegenden Bereichsschiebers wäre, dass beide thumbs (?) unabhängig bewegt werden könnten, Bewegung des tmin thumbs würde den tmax thumbs so mitbewegen lassen, dass die Länge der Zeitperiode gleich bleibt. Nur eine Bewegung des tmax thumbs kann die Länge der Zeitperiode verändern. Das ist deswegen so gemacht, weil der User meistens interessiert ist Zeitperioden gleicher Dauer zu vergleichen.
Daten Übersicht, die der Zeit Schieber bereitstellt:

Verwendung des Sliders (Schiebers) um einen Zeitabschnitt zu definieren:
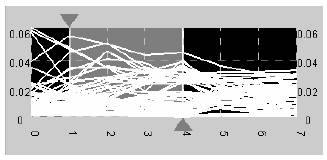
Wenn der Zeitschieber nun verwendet wurde, können in Folge Standard-Mehrbereichs (Multi-range) Schieber verwendet werden um gewünschte Bereiche von Werten bzw. Wertveränderungen zu wählen. So wie in existierenden visuellen Abfragetools, wird dann der anfängliche Überblick durch das gefilterte Subset nach der Abfrageformulierung ersetzt.
In der folgenden Abbildung ist die Wertabfragerepräsentation, überlagert auf das entsprechende gefilterte Ergebnisset.
Graphischer Zeitschieber verwendet um eine Wertbereichabfrage zu machen:
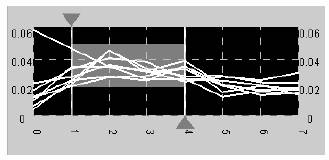
Um Prozentsätze in Bezug auf den Maximalwert und Fold Veränderung abzufragen, kann der passende Reskalierungsmodus mittels der Menübar ausgewählt werden und die Daten werden entsprechend reskaliert. Dieser Ansatz erfüllt die User Anforderungen, dass sie zwischen verschiedenen Reskalierungen wechseln können um ihre Ergebnisse zu verifizieren. Da es zu verwirrend für die Benützer wäre, werden nicht mehrere Reskalierungen gleichzeitig angezeigt.
Durch die Kombination Zeitschieber und Mehrbereichs Abfrage parameter schieber können alle gewünschten Abfragen durchgeführt werden. Die Abfragresultate werden als Wert/Zeit Graphiken dargestellt.
Eine wesentliche Beschränkung dieser Kombination ist jedoch, dass keine visuellen Abfragen über Wertveränderungen gemacht werden können. Daher gibt es auch Detailansichten um die Übersicht zu vervollständigen.
3.2 Detailansicht
In der traditionellen Wert gegen Zeit Darstellung der Zeitreihen, sind die Merkmale abhängig von einer einzelnen Zeit. Zusätzlich werden jedoch andere Ansichten der Daten benötigt, die Merkmale der Daten, abhängig von einer Zeitperiode zeigen. Diese Merkmale sind Veränderung und fold change. Diese Attribute sind in einer Detailansicht als Streudiagramme (scatter plots) dargestellt, wie in der Abbildung dargestellt:
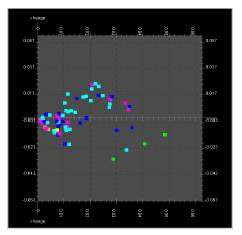
Die Y-Achse sind entweder Veränderung oder Fold-Veränderung abhängig vom Reskalierungsmodus und die X-Achse sind die durchschnittlichen Werte. Die druchschnittlichen Werte sind für jedes einzelne Gen für die Zeitperiode berechnet worden und nicht zu verwechseln mit dem Mittelwert aller Gene in dem Zeitabschnitt. Während eine messbare Anzeige von Expressionsveränderung dem User erlaubt Abfragen entsprechend steigender oder abfallender Expression zu machen, so erlaubt eine messbare Anzeige der durchschnittlichen Wertes dem User abzufragen, ob eine Gruppe von Genen aktiv/nicht aktiv ist. Ein weiterer Vorteil des Scatter plots der ausgewählten Zeitperiode ist auch, dass die Repräsentation einzelner Punkte geeignet ist, funktionelle Klassifikationen durch Farbkodierung zu überlagern. Falls es mehr funktionelle Klassifikationen als unterscheidbare Farben gibt, kann die Farbkodierung abgeschaltet werden und User kann einzelne Gruppen hervorheben indem er auf deren Namen in der Gruppierungsschaltfläche klickt.
Veränderungs und Durchschnittswertabfragen können durchgeführt werden, indem man eine Zelle (Box) wegzieht (dragging?), welche den erwünschten Bereich der mittleren Werte bzw. der Veränderung der Werte definiert. Wenn einmal eine Abfrage formuliert wurde, kann sie durch Clicking und Dragging der Ecken/Kanten der visuellen Abfrage verändert werden. Abfrageergebnisse werden in der Ansicht aller Daten hervorgehoben (highlighted), so dass der User die Abfrage mit demselben Überblick, die in der anfänglichen Formel verwendet wurde, modifizieren kann. Das sieht an in der Abbildung (die gezeigte Abfrage wählt eine Gruppe von Genen mit hoher jedoch fallender Expression):
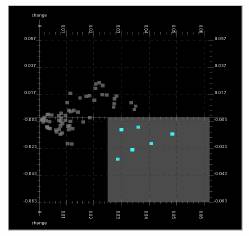
3.3. Koordination der Ansichten
Wenn der Scatter Plot (das Streudiagramm) verwendet wird um die gesamte beobachtete Zeitspanne darzustellen, ist es en rudimentärer Überblick über die Daten. Falls ein Zeitabschnitt ausgewählt wird, so ist es ein Überblick über diesen Zeitabschnitt. Die wahre Macht der Visualisierung erlangt man, wenn mehrere Ansichten zusammen verwendet werden um die Daten interaktiv zu erforschen.
Diese Funktionalität wird unterstützt durch verschiedene Grade der Koordination zwischen den Ansichten.
Ein Weg die Diagramm und die Scatterplot Ansicht zu koordinieren ist, dass wenn der User eine Zeitperiode in der Diagrammansicht festsetzt, die Merkmale dieser Zeitperiode in der Scatterplot Ansicht angezeigt werden.
Da es notwendig sein könnte zu zeigen, wie sich die Zeitreihe in aufeinanderfolgenden Zeitperioden entwickelt und wie die Verbindung zwischen den Positionen der Repräsentationen unterschiedlicher Zeitperioden ist, werden die abgeleiteten Parameter im Scatterplot als interpolierte Werte berechnet. Die Thumbs des sliders können viel genauer (less granularity?) als die ursprünglichen Zeitreihen bewegt werden, die Genrepräsentationen im Scatterplot display bewegen sich in kleineren Schritten. Das macht es für den User möglich , die Scatterplot Ansicht zu animieren um einen Eindruck zu gewinnen, wie sich die Expression für einzelne bzw. für Gruppen von Genen über die Zeit verändert. Direkte Manipulation der festgesetzten Zeitperiode erlaubt es hier dem User die einzelnen Frames einfach zu vergleichen, was bei traditionellen Animationen nicht möglich ist.
Lineare Interpolation ist generell nicht geeignet für microarray Daten, hier wird sie aber nur zu dem Zweck verwendet, dass der User verschiedene Genrepräsentationen miteinander in Verbindung setzten, während er die Zeitperiode mittels dem Zeitschieber anpasst.
Wenn der User auf Genrepräsentationen in der Scatterplot Ansicht oder in der Genliste klickt, werden sie im Scatterplot markiert und in der Diagramm (Kurven-) Ansicht markiert/hervorgehoben. Das ermöglicht dem User die Scatterplot Ansicht mit Hilfe der vertrauten Diagrammansicht besser zu verstehen. Auch die Abfrageergebnisse werden auf ähnliche Weise koordiniert. Wenn eine Abfrage formuliert wird, werden die Resultate einerseits durch Ersetzung im Diagramm angezeigt, andererseits durch Hervorhebung/Markierung im Scatter Plot. Die Abfragen werden inkrementell angepasst, somit wird das Ergebnis im Display schnell upgedatet.
Koordination der Ansichten ist auch nötig um verschiedene Abfragen zu kombinieren um Ähnlichkeitsmuster aufzudecken. Währende der Formulierung und Modifikation einer einzelnen Abfrage kann der User inkrementell die Abfrageparameter anpassen indem er die slider thumbs oder die visuellen Abfragekomponenten manipuliert. Um eine Abfrage hinzuzufügen, drückt der User den add button (+).
Wenn eine neue Abfrage hinzugefügt wird, hat sie nur eine Zeitperiode und einen REskalierungsmodus. Diese Parameter filtern nicht bestehende Abfragen. Zum Beispiel ist es möglich, bei einer bestehenden Abfrage die Reskalierung und die festgesetzte Zeitperiode zu ändern. Wenn ein User Gene mit einer früh beginnenden steigenden Expression in Logskalierung in einer anfänglichen Abfrage selektiert, so kann er nun durch hinzufügen einer neuen Abfrage, die Zeitperiode ändern um zu sehen, wie diese Gene sich in einer späteren Periode verändern.
Auch kann er nach dem er eine Abfrage spezifiziert hat in den nicht-rresklaiert Modus wechseln um zu sehehn, ob z.b. Gene eine ansteigende log-skalierte Expression haben aufgrund einer Verstärkung unwichtiger minimaler Variationen. Unabhängig davon, welche Abfrage ausgewählt ist, entspricht die markierte Auswahl immer den Originaldaten gefiltert mit alle Abfragen. Klicken zwischen den verschiedenen Abfragen ohne Änderung der Abfragen, ändert nur die visuelle Abfragerepräsentation bzw. die Zeitperiode oder Sklaierung falls passend.
In der Query Kombinationsschaltfläche sind die Abfragen textuell dargestellt mit den Abfrageparametern, den Zeitabschnittkontext und die Reskalierung. (Rot umrandet, falls gerade ausgewählt)
Mit dem remove Button (-) können Abfragen entfernt werden.
Schlussendlich kann der User Abfragen und Ergebnisse im Menü speichern und wieder öffnen.